Virología
Mecanismos de acción de compuestos antivirales aislados en plantas: el Virus de la Inmunodeficiencia Humana como modelo.
Mecanismos de replicación del VIH
Antes de discutir los trabajos relacionados con las propiedades anti-VIH de algunos compuestos naturales, es importante comprender los pasos del ciclo de multiplicación del virus, ya que ello permitirá entender cómo y donde actúan estas sustancias.
El VIH pertenece al género lentivirus de la familia Retroviridae. Los viriones maduros miden entre 100 y 120 nm de diámetro y presentan una membrana lipídica que rodea una nucleocápside de forma cónica en la que se encuentra el genoma viral y ciertas proteínas virales (Figura 1): la retrotranscriptasa, la proteasa, la integrasa y las proteínas Vif, Vpr, Nef, Vpu (en el caso del VIH de tipo 1) y Vpx (en el caso del VIH-2) (Luciw, 1996. Sierra et al, 2005). También se encuentran ciertos factores celulares, en particular a nivel de la membrana del virión que es adquirida cuando la partícula viral gema a través de la membrana plasmática de la célula durante le última fase de la infección (Sierra et al, 2005).
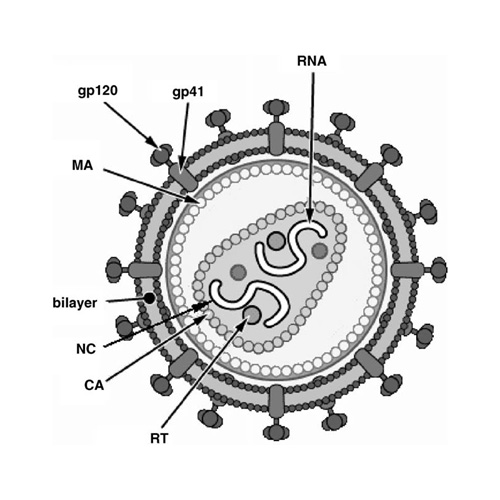
Figura 1: Diagrama de viriones de VIH maduros. Se muestran las proteínas superficiales del virión (gp 120 y gp 41), la bicapa lipídica, las proteínas de la matriz (MA) que forman una cubierta alrededor de la nucleocáspside cónica. Esta última está constituida por las proteínas de la cápside (CA) y protege las moléculas de RNA que constituyen el genoma viral. Además contiene numerosas proteínas virales entre las que se encuentran las de la nucleocápside (NC) y las retrotranscriptasas (RT) (Tomado de Sierra et al, 2005).
El genoma está constituido por dos moléculas idénticas de RNA de simple cadena de 9.2 kb, con polaridad positiva y que sirven de templado para la retrotranscriptasa viral. Esta enzima transforma el RNA viral de simple cadena en DNA de doble cadena que luego se incorpora al genoma del hospedador. La forma viral integrada y latente se denomina provirus. El genoma proviral presenta una región codificante central en la que se encuentran los genes virales, algunos de los cuales se encuentran solapados (Figura 2) (Luciw, 1996).
Figura 2: Genomas del VIH-1 y 2. Las regiones codificantes se encuentran flanqueadas por secuencias de DNA redundantes denominadas LTR (Long Terminal Repeats) que intervienen en la regulación de la expresión génica (Tomado de Weiss, 2000).
Flanqueando a la región codificante se encuentran secuencias de DNA redundantes (idénticas tanto en el extremo 3' como en el extremo 5') denominadas LTRs (del inglés "Long Terminal Repeats"). Las LTRs son regiones reguladoras de la expresión génica y se encuentran divididas en tres dominios con distintas funciones (U3, R y U5) (Coffin, 1996). El dominio U3 contiene elementos promotores de la transcripción y secuencias reconocidas por otros factores celulares que modulan la transcripción al influir en la tasa de iniciación. El dominio R presenta el elemento de respuesta de activación en trans (Tar, del inglés "trans-activation response element"). Tar es una secuencia nucleotídica que se encuentra tanto en el RNA como el DNA viral formando un bucle que es reconocido por la proteína viral Tat. Ésta controla la expresión de los genes virales (Luciw, 1996. Coffin, 1996).
La transcripción se inicia en el límite entre la región U3/R y los transcritos que se producen tienen las modificaciones características de los RNAm eucarióticos: un extremo 5' poliadenilado y una caperuza de 7-metil-guanosina en el extremo 3' (Cann, 2001). Los genes virales y la función de sus respectivos productos aparecen señalados en la Tabla 2. La infección con el VIH comienza cuando los viriones se unen a las células mediante la interacción entre la glicoproteína gp120, presente en la cubierta viral, y el receptor celular CD4 situado en la membrana plasmática de macrófagos y linfocitos T colaboradores, aunque se han encontrado receptores alternativos como la galactosil ceramida en células CD4- (Luciw, 1996. Sierra et al, 2005). La unión de la gp120 al receptor CD4 causa un cambio conformacional en la glicoproteína exponiendo una región que se une a los co-receptores celulares CCR5 y CXCR4. Éstos determinan el tropismo de las cepas de VIH: las que utilizan el co-receptor CXCR4 infectan los linfocitos T (T trópicas), mientras que las que interaccionan con CCR5 infectan a macrófagos y monocitos (M-trópicas) (Princen y Schols, 2005). Después de la unión a los co-receptores, la membrana viral se fusiona con la membrana celular y la nucleocápside es liberada al citoplasma (Figura 3) (Luciw, 1996).
Tabla 2. Nomenclatura, función y localización de las proteínas codificadas por el genoma viral
|
Gen |
Proteína |
Función |
Localización |
|
Gag |
Pr55 gag |
Dirige el ensamblaje de los viriones; poliproteína precursora de las proteínas de la nucleocápside: matríz (MA o p17), cápside (CA o p24), nucleocápside (NC o p9) y p7 |
Nucleocápside del virión |
|
Pol |
Pr 160 gag-pol |
Poliproteína precursora de la enzimas virales: proteasa (PR o p10), retrotranscriptasa o RNasa-H (RT o p51/p66) e integrasa (IN o p32) |
Virión |
|
Vif |
P23 |
Interviene en la decapsidación del virión; limita el efecto antiretroviral de la proteína celular APOBEC3G al favorecer su degradación; participa en la decapsidación |
Citoplasma celular |
|
Vpx (solo presente en VIH-2) |
P16 |
Permite que el DNA viral llegue al núcleo celular |
Virión |
|
Vpr |
P15 |
Ayuda a detener el ciclo celular y evitar la proliferación de las células; permite que el DNA viral llegue el núcleo celular |
Virión |
|
Tat |
P 14 |
Transactivador transcripicional: se une a TAR y a factores celulares promoviendo la elongación y la formación de complejos de iniciación |
En el núcleo celular |
|
Rev |
P 19 |
Transcativador post-transcripcional: se une a RRE y a factores celulares regulando el "splicing", el transporte al citoplasma y la traducción de los transcritos. |
En el núcleo celular |
|
Vpu (solo presente en VIH-1) |
P 16 |
Induce la degradación de la moléculas de CD4; forma poros transportadores de iones que incrementan la liberación de virus |
Proteína de membrana celular |
|
Env |
Gp 160 |
Secuestra las moléculas de CD4 a nivel del retículo endoplasmático; poliproteína precursora de las proteínas de le envoltura:
- Superficie (SU o gp120): Proteína de unión al receptor CD4
- Transmembrana (TM o gp41): Proteína de fusión a la membrana
|
Envoltura del virión y en la membrana plasmática |
|
Nef |
P 27 |
Acelera la endocitosis y subsecuente degradación de las moléculas de CD4 así como las moléculas del MHC de tipo I y II; interviene en la decapsidación; favorece la transcripción mediada por el intensificador celular NF-κB |
Citoplasma, membrana plasmática |
(Adaptado de Luciw, 1996 y Sierra et al, 2005).
Una vez en el citoplasma, ocurre la decapsidación de la nucleocápside viral mediante la acción de factores celulares y de las proteínas virales MA, Nef y Vif (Sierra et al, 2005). El RNA viral que se libera se retrotranscribe generando DNA de doble cadena. Este proceso requiere de un cebador constituido por una molécula de tRNAlys que se encuentra ubicada cerca del extremo 5' del RNA viral unida a una secuencia denominada PBS (Primer Binding Site) (Luciw, 1996).
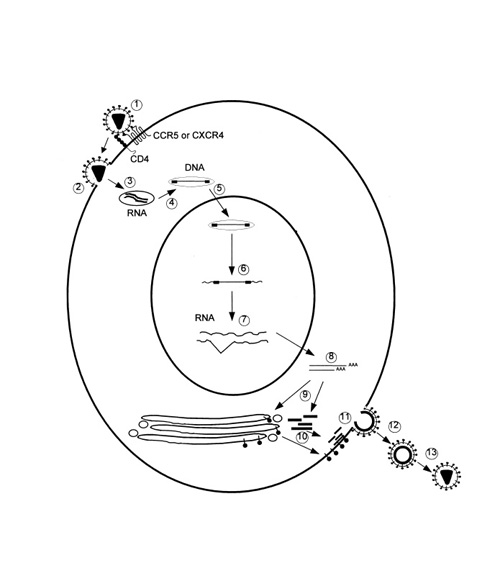
Figura 3. Ciclo de replicación del VIH. Se muestran las diferentes etapas del ciclo. 1) Adsorción, 2) Fusión, 3) Entrada, 4) Transcripción reversa, 5) Transporte al núcleo, 6) Integración cromosomal del genoma proviral, 7) Transcripción del RNA, 8) Exportación nuclear del RNA, 9) Traducción y procesamiento, 10) Transporte a la membrana, 11) Ensamblaje, 12) Gemación, 13) Maduración (Tomado de Weiss, 2000).
La primera cadena de DNA se extiende hacia el extremo 5' del RNA viral generando un pequeño fragmento que es complementario al dominio R del genoma viral (Cann, 2001). La actividad RNAsa H de la RT degrada el complejo RNA/DNA y libera el DNA recién sintetizado que se une al domino R del extremo 3' del RNA viral (Figura 4). Esta es la primera transferencia de hebra y viene seguida por la síntesis integral de la primera cadena de DNA. La segunda hebra de DNA requiere de la presencia de dos cebadores situados en la cadena de DNA que sirve de templado: dos motivos polipurínicos (PPT) situados en el límite del dominio U3 y al final del gen pol (cPPT). La síntesis de DNA que se inicia en el límite de U3 genera un fragmento que se une al extremo 3' del DNA molde por complementariedad y que se sigue extendiendo hacia el extremo 5' del mismo por la acción de la RT (Figura 4). Simultáneamente, se lleva a cabo la síntesis de DNA desde cPPT hacia el extremo 5' del DNA molde, de manera tal que se generan dos fragmentos de DNA con una secuencia repetida de alrededor de 100 nucleótidos. Esta secuencia es desplazada, escindida y los dos fragmentos de DNA son ligados por la acción de enzimas celulares, generando el DNA linear de doble cadena (Luciw, 1996. Cann, 2001).
La retrotranscripción es el principal proceso responsable de la diversidad antigénica del virus ya que la retrotranscriptasa carece de la actividad exonucleasa 3'-5', presente en las DNA polimerasas celulares, por lo que la tasa de mutaciones es muy superior a la que se registra en células eucariotas. Se estima que el genoma viral experimenta de una a tres mutaciones por ciclo de replicación (Luciw, 1996).
Figura 4: Mecanismo de la retrotranscripción. Ver detalles en el texto (Tomado de Coffin, 1996).
Una vez sintetizado el DNA viral, éste se dirige al núcleo mediante la acción de Vpr, una proteína que ancla el DNA viral a la membrana nuclear permitiendo que penetre al núcleo a través de los poros nucleares (Sierra et al, 2005). El DNA es incorporado al genoma del hospedador mediante la acción de la integrasa viral que realiza cortes escalados en los genomas celular y viral, uniéndolos y rellenando los espacios dejados por las bases no pareadas (Cann, 2001). Las primeras rondas de transcripción llevadas a cabo por la RNA polimerasa II celular sobre el DNA viral integrado, generan varios transcritos que migran al citoplasma mediante una vía independiente de Rev. Durante esta fase de replicación temprana se producen múltiples cortes y empalmes de los RNAm y sólo las proteínas regulatorias Tat, Nef y Rev se expresan. Al alcanzar niveles suficientes, Tat se une a Tar y controla la transcripción de los genes virales. Cuando Rev alcanza niveles adecuados, se une a una estructura de los transcritos denominada elemento de respuesta a Rev (RRE) facilitando su paso a través de la membrana nuclear y protegiéndolos del ?splicing?, de manera que se generan RNAm sin cortes o con un único corte que producen las otras proteínas virales y el RNA genómico (Sierra et al, 2005). El gen env, que codifica para las proteínas de unión a CD4 (gp120) y para las proteínas virales transmembranales (gp41), se expresa inicialmente como una poliproteína precursora denominada gp160. Ésta es glicosilada dentro del retículo endoplasmático y el aparato de Golgi, en donde una endoproteasa escinde la proteína para generar gp120 y gp41 (Luciw, 1996). Estas proteínas son transportadas por vesículas celulares hasta la membrana plasmática. Los genes gag-pol se traducen para generar la poliproteínas Gag y Gag-pol que son las precursoras de las 6 proteínas estructurales codificadas por gag y las proteasas, integrasas y retrotranscriptasas codificadas por pol. Estas poliproteínas son procesadas proteolíticamente durante la maduración (Sierra et al, 2005). Después de la traducción, las proteínas Gag y Gag-pol migran hacia la membrana celular y empiezan a ensamblarse dirigidas por Gag. Debido a que las proteínas virales se expresan sobre la superficie celular, se puede producir la fusión entre células infectadas y células sanas que expresen el receptor CD4 generando células gigantes multinucleadas (sincitios). La formación de sincitios constituye uno de los mecanismos de propagación del virus y es parcialmente responsable de la disminución progresiva del número de linfocitos T característico del SIDA (Geleziunas y Greene, 2000). Durante el ensamblaje de las partículas virales se asocian las enzimas virales, el genoma viral, el cebador celular RNAtlys y otros compuestos celulares. Los viriones inmaduros geman a través de la membrana plasmática, lo que activa la proteasa viral que escinde las poliproteínas Gag y Gag-pol, liberando las proteínas estructurales y las enzimas (Sierra et al, 2005). Todas ellas interactúan con las proteínas de la cápside (CA) y de la nucleocápside (NC) para generar los viriones maduros. El proceso de ensamblaje y gemación requiere de la disminución del número de moléculas CD4 celulares para evitar su interacción con las proteínas gp120 recién sintetizadas. En este proceso intervienen las proteínas Env, Nef y Vpu. La primera se une a las moléculas de CD4 que pasan por el retículo endoplasmático durante su maduración, mientras que la segunda acelera la endocitosis y la degradación subsecuente de CD4. La proteína Vpu produce canales iónicos en la membrana plasmática que incrementan la tasa de liberación de virus (Luciw, 1996. Sierra et al, 2005).
Una vez conocido en detalle el mecanismo molecular mediante el cual el virus lleva a cabo su multiplicación, muchos investigadores se han enfocado en desarrollar terapias que bloqueen algunos de los pasos antes descritos. En particular, ciertos grupos de investigación han centralizado su interés en estudiar como actúan los diferentes compuestos naturales con propiedades anti-VIH aislados de plantas.
|